Baza podataka orijentacija proteina u membranama
Baza podataka orijentacija proteina u membranama (engl. Orientations of Proteins in Membranes, OPM) sadrži prostorne orijentacije proteinskih tri-dimenzionalnih struktura u odnosu na lipidni dvosloj.[1][2][3] Ova baza podatak je bila korišćena u eksperimentalnim i teoretskim studijama za membranu-vezanih proteina.[4][5][6][7][8]
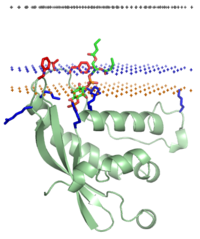
Opis
urediProteinske strukture su uzet iz baze podataka proteina. Pozicije proteina u hidrofobnoj ploči su izračunate koristeći model implicitne solvatacije.
OPM sadrži strukturnu klasifikaciju za membranu-vezanih proteina u familije i superfamilije (baziranu na SCOP klasifikacije), membransku topologiju, i tip odredišta membrane za svaki protein. Svi fajlovi sa proteinskim koordinatama sa izračunatim membranskim granicama su slobodno dostupni.
Ovaj sajt omogućava vizuelizaciju proteinskih struktura sa ravnima membranskih granica koristeći Jmol, MDL Chime i WebMol.
Prednosti
urediOva baza podataka sadrži prostorne pozicije transmembranskih i perifernih membranskih proteina u lipidnom dvosloju, zajedno sa bazom podataka transmembranskih proteina[9] koja uvrštava samo transmembranske proteine. Izračunate pozicije proteina su bile kompajlirane sa relevantnim eksperimentalnim podacima za 24 transmembranska i 53 periferno membranska proteina[3] uključujući mesto-usmereno spinsko obeležavanje,[10] hemijsko obeležavanje, merenja membranskih vezivnih afiniteta proteinskih mutanata,[11] fluorescentnu spektroskopiju,[12] NMR spektroskopiju rastvora ili čvrstog-stanja ,[13] ATR FTIR spektroskopiju,[14] i difrakcione studije proteina u lipidnim slojevima X-zracima ili neutronima[15].
Nedostaci
urediOPM kolekcija perifernih membranskih proteina nije kompletna. Ova baza podataka verovatno sadrži manje od 50% PDB perifernih proteina, zato što za-membranu-učvršćujući elementi perifernih proteina (amfifilni alfa heliksi, izloženi nonpolarni ostaci, ili lipid-vezani proteini) nedostaju ili su neuređeni u eksperimentalnim proteinskim strukturama, i stoga mod protein-membranske veze ne može biti računski predviđen[3].
Vidi još
urediLiteratura
uredi- ^ Lomize, Mikhail A.; Lomize, Andrei L; Pogozheva, Irina D.; Mosberg, Henry I. (2006). „OPM: Orientations of Proteins in Membranes database” (PDF). Bioinformatics. 22 (5): 623—625. PMID 16397007. doi:10.1093/bioinformatics/btk023.
- ^ Lomize, Andrei L; Pogozheva, Irina D.; Lomize, Mikhail A.; Mosberg, Henry I. (2006). „Positioning of proteins in membranes: A computational approach” (PDF). Protein Science. 15 (6): 1318—1333. PMC 2242528 . PMID 16731967. doi:10.1110/ps.062126106.
- ^ а б в Lomize, Andrei L; Pogozheva, Irina D.; Lomize, Mikhail A.; Mosberg, Henry I. (2007). „The role of hydrophobic interactions in positioning of peripheral proteins in membranes” (PDF). BMC Structural Biology. 7 (44): 44. PMC 1934363 . PMID 17603894. doi:10.1186/1472-6807-7-44.
- ^ Park, Yungki; Helms, Volkhard (2007). „On the derivation of propensity scales for predicting exposed transmembrane residues of helical membrane proteins”. Bioinformatics. 23 (6): 701—708. PMID 17237049. doi:10.1093/bioinformatics/btl653.
- ^ Marsh, Derek; Jost, Micha; Peggion, Cristina; Toniolo, Claudio (2007). „TOAC spin labels in the backbone of alamethicin: EPR studies in lipid membranes”. Biophysical Journal. 92 (2): 473—481. PMC 1751395 . PMID 17056731. doi:10.1529/biophysj.106.092775.
- ^ Punta, Marco; Forrest, Lucy R.; Bigelow, Henry; Kernytsky, Andrew; Liu, Jinfeng; Rost, Burkhard (2007). „Membrane protein prediction methods”. Methods. 41 (4): 460—474. PMC 1934899 . PMID 17367718. doi:10.1016/j.ymeth.2006.07.026.
- ^ Cherezov, V; Yamashita, E; Liu, W; Zhalnina, M; Cramer, WA; Caffrey, M (2006). „In Meso Structure of the Cobalamin Transporter, BtuB, at 1.95 Ångstrom Resolution”. J. Mol. Biol. 364 (4): 716—734. PMC 1808586 . PMID 17028020. doi:10.1016/j.jmb.2006.09.022.
- ^ Páli, Tibor; Bashtovyy, Denys; Marsh, Derek (2006). „Stoichiometry of lipid interactions with transmembrane proteins - Deduced from the 3D structures”. Protein Sci. 15 (5): 1153—1161. PMC 2242517 . PMID 16641489. doi:10.1110/ps.052021406.
- ^ „Protein Data Bank of Transmembrane Proteins”. Budapest, Hungary: Institute of Enzymology. Архивирано из оригинала 25. 12. 2013. г. Приступљено 12. 10. 2010.
- ^ Malmberg, Nathan J.; Falke, Joseph J. (2005). „Use of EPR power saturation to analyze the membrane-docking geometries of peripheral proteins: applications to C2 domains”. Annu Rev Biophys Biomol Struct. 34: 71—90. PMID 15869384. doi:10.1146/annurev.biophys.34.040204.144534.
- ^ Spencer, Andrew G.; Thuresson, Elizabeth; Otto, James C.; Song, Inseok; Smith, Tim; DeWitt, David L.; Garavito, R. Michael; Smith, William L. (1999). „The membrane binding domains of prostaglandin endoperoxide H synthases 1 and 2. Peptide mapping and mutational analysis”. J Biol Chem. 274 (46): 32936—42. PMID 10551860. doi:10.1074/jbc.274.46.32936.
- ^ Lathrop, Brian; Gadd, Martha; Biltonen, Rodney L.; Rule, Gordon S. (2001). „Changes in Ca2+ affinity upon activation of Agkistrodon piscivorus piscivorus phospholipase A2”. Biochemistry. 40 (11): 3264—72. PMID 11258945. doi:10.1021/bi001901n.
- ^ Kutateladze, Tatiana; Overduin, Michael (2001). „Structural Mechanism of Endosome Docking by the FYVE Domain”. Science. 291 (5509): 1793—6. PMID 11230696. doi:10.1126/science.291.5509.1793.
- ^ Tatulian, Suren A.; Qin, Shan; Pande, Abhay H.; He, Xiaomei (2005). „Positioning membrane proteins by novel protein engineering and biophysical approaches”. J Mol Biol. 351 (5): 939—947. PMID 16055150. doi:10.1016/j.jmb.2005.06.080.
- ^ Hristova, Kalina; Wimley, William C.; Mishra, Vinod K.; Anantharamiah, G.M.; Segrest, Jere P.; White, Stephen H. (1999). „An amphipathic α-helix at a membrane interface: a structural study using a novel X-ray diffraction method”. J Mol Biol. 290 (1): 99—117. PMID 10388560. doi:10.1006/jmbi.1999.2840.